A partir da década de 1970, ficou mais fácil analisar a molécula de DNA
com o isolamento da enzimas de restrição. Estas enzimas são endonucleases, ou seja, no interior (daí o prefixo endo- dentro) das moléculas de DNA, cortando-as em locais bem definidos.
São
enzimas produzidas normalmente por bactérias e que possuem a
propriedade de defendê-las de vírus invasores. Essas substâncias
“picotam” a molécula de DNA sempre em determinados pontos, levando a
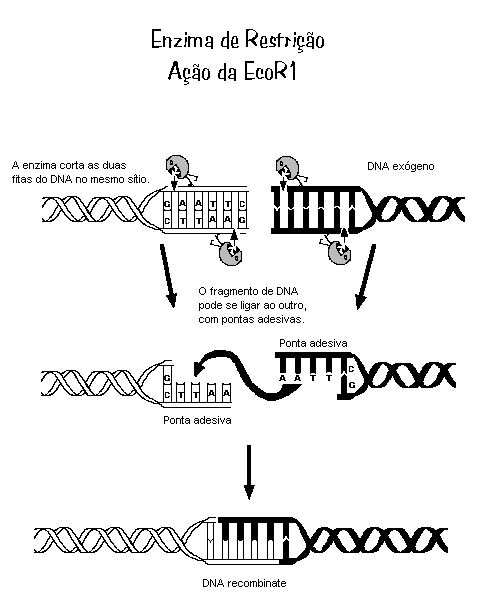
produção de fragmentos contendo pontas adesivas, que
podem se ligar a outras pontas de moléculas de DNA que tenham sido
cortadas com a mesma enzima. Em Engenharia Genética, a obtenção dos
fragmentos de DNA serve para criar, in vitro (em tubo de ensaio ou no laboratório), novas moléculas, recortando e colando vários pedaços de informações.
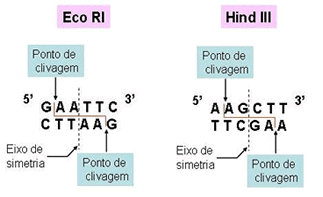
Uma das primeiras enzimas de restrição a ser isolada foi a EcoRI, produzida pela bactéria Escherichia coli. Essa enzima reconhece apenas a sequência GAATTC e atua sempre entre o G e o primeiro A.
O local do “corte”, o local de uma enzima, é
conhecido como sítio alvo. Você pode perguntar por que essa enzima não
atua no DNA da própria bactéria? Isso não ocorre devido à existência de
outras enzimas protetoras, que impedem a ação das enzimas de restrição
no material genético da bactéria.
As enzimas de restrição reconhecem e atuam sobre sequências específicas de DNA,
catalisando a destruição de uma ligação fosfodiéster entre dois
nucleotídeos consecutivos ligados a determinadas bases. Os nucleotídeos
entre os quais a enzima corta, ou seja, entre os quais promove a
hidrólise, encontram-se no interior dessas mesmas sequências específicas
de reconhecimento (ver imagem).
Cada molécula de DNA pode ser composta de várias repetições da sequência
GAATTC ao longo de toda sua extensão. Portanto ao contato com a ezima
EcoRI a fita de DNA pode ser clivada "cortada" em diversos lugares,
gerando vários pedaços, de tamanhos diferentes.
Fonte: http://www.sobiologia.com.br

Nenhum comentário:
Postar um comentário